Um wirksame Medikamente gegen Viren zu identifizieren, müssen Wissenschaftler zunächst verstehen, wie sich diese Krankheitserreger vermehren. Nur dann können sie deren Reproduktionsmechanismus mit effektiven Wirkstoffen außer Kraft setzen. Mit neuen Modellrechnungen sind Berliner Forscher jetzt dem Verständnis dieses Mechanismus einen Schritt näher gekommen. Anhand der Helikase des Hepatitis-C-Virus simulierten sie, wie dieses Enzym die Doppelhelixstruktur des viralen Erbguts aufbricht und dieses so für die Reproduktion aufbereitet.
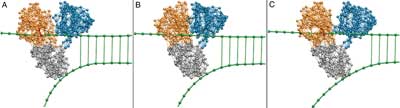
Wie eine Raupe kriecht die Hepatitis-C-Helikase dabei über die DNA und trennt die beiden Stränge des Erbgut-Moleküls. Zwei Module des Enzyms heften sich an den einen Strang und schieben sich abwechselnd jeweils um eine Base vorwärts. Das dritte Modul drückt dabei die beiden Stränge auseinander, so die Forscher vom Fritz-Haber-Institut der Max-Planck-Gesellschaft in Berlin in der Fachzeitschrift „Proceedings of the National Academy of Sciences“ (PNAS).
Ohne Helikasen keine Vermehrung
Ohne Helikasen könnten Viren sich nicht vermehren. Sie nutzen diese Enzyme, um ihre eigene RNA oder die DNA der Wirtszelle aufzuspalten – einer der wichtigsten Schritte, um ein neues Virus herzustellen. Nach welchem Muster die Helikase dabei etwa im Hepatitis-C-Virus vorgeht, konnten Wissenschaftler bisher nur erahnen – ihre Struktur ist zu komplex, um sie mit Mikroskopen detailliert zu beobachten. Daher entwickeln Biophysiker Computermodelle, um die komplexen Mechanismen der Virenreproduktion zu verstehen.
Arbeitszyklus des Helikase-Enzyms enträtselt
Holger Flechsig und Alexander Mikhailov vom Fritz-Haber-Institut der Max-Planck-Gesellschaft haben mit einem neuen Modell simuliert, wie die Helikase des Hepatitis-C-Virus die Helixstruktur seines Erbguts aufspaltet. Für ihre Untersuchung nutzten sie ein Modell aus Hepatitis-C-Helikase und DNA.