Allerdings ist es angesichts der Fülle von Medikamenten und Mutationen schwer, aus diesen Tabellen die geeigneten Arzneien für eine neue Therapie abzuleiten. Das Decision-Tree-Verfahren automatisiert diesen Entscheidungsprozess. Wie bei der Support-Vektor-Maschine (SVM) auch geht es hier darum, die Virensequenz aus einer Blutprobe des Patienten einer von zwei Klassen zuzuordnen – „resistent“ und „nicht resistent/suszeptibel“.
In der Trainingsphase füttern die Forscher den Algorithmus wiederum mit den Aminosäuresequenzen aus den Viren der 900 Referenzpatienten. In diesem Fall ordnen sie den Sequenzen jedoch keine genauen Resistenzfaktoren, sondern lediglich die Information „resistent“ oder „suszeptibel“ zu. Der Computer setzt dann alle 900 Sequenzen zueinander in Beziehung und zieht selbstständig logische Verknüpfungen zwischen den verschiedenen Mutationen.
Computer ermittelt Resistenzrisiko
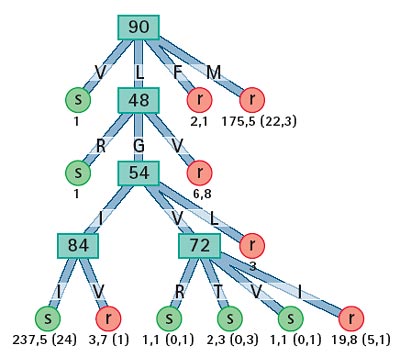
Füttert man den Decision-Tree-Algorithmus dann mit einer Patientensequenz, gleicht der Computer die Daten mit dem erlernten Wissen ab. Er weiß, bei welcher Kombination von Mutationen Resistenzen gegen ein Medikament auftreten. So liefert er einen Entscheidungsbaum, in dem man wie in einem Flussdiagramm von Schritt zu Schritt nachvollziehen kann, bei welcher Abfolge von Mutationen eine Resistenz vorliegt. Etwa so: „Befindet sich an Position 90 die Aminosäure Valin, ist das Virus nicht gegen das Medikament Saquinavir resistent. Befindet sich an Position 90 die Aminosäure Valin und an Position 48 ebenfalls die Aminosäure Valin, dann ist das Virus resistent.“
Je mehr Stationen der Decision Tree hat, desto komplexer wird das Beziehungsgeflecht. Desto deutlicher wird auch, wie schwer es ist, die Zusammenhänge allein mit Menschenverstand entwirren zu wollen. In Geno2Pheno setzen sich die SVM und der Decision-Tree-Algorithmus gleichzeitig in Bewegung, wenn man eine Sequenz eintippt. Die Ergebnisse zeigt der Computer dann in einer gemeinsamen Tabelle an.
„Inzwischen verschreiben wir keine neue HAART-Kombination mehr, ohne uns mit Geno2Pheno abzusichern“, sagt Tillmann Schumacher, der seit 20 Jahren HIV-Positive betreut. In den frühen 1990er-Jahren, erinnert sich der Oberarzt der Inneren Abteilung des Kölner Krankenhauses der Augustinerinnen, gab es erst wenige Medikamente. Ein erfahrener Behandler konnte recht gut einschätzen, welche Medikamente er verabreichen sollte. Heute sei die Situation wesentlich komplexer. Man müsse strategisch vorgehen, um Kreuzresistenzen zu vermeiden und eine Therapie möglichst lange erfolgreich betreiben zu können. Die Software aus Saarbrücken sei eine gute Hilfe. Die Erfahrung des Arztes ersetze sie allerdings nicht.
Der stetige Kampf gegen die Viruslast
Denn zu berücksichtigen sind stets auch die Nebenwirkungen der Medikamente. Darüber hinaus müssen die Arzneimittel – zehn bis 20 Tabletten am Tag – diszipliniert und auf die Stunde genau eingenommen werden. Je nachdem, welchen Tagesrhythmus ein Patient hat, kommen andere Präparate in Frage: Tabletten, die man vor oder nach dem Essen, morgens oder abends, mit oder ohne Nahrung einnimmt. So scheiden manche Wirkstoffe aus, obwohl Geno2Pheno sie als geeignet identifiziert hat.
{3r}
„Man muss ungeheuer diszipliniert sein, um stets an die Medikamente zu denken“, sagt Thomas Fenkl. „Etwa einmal im Monat geht mir eine Dosis durch die Lappen. Das sehe ich dann oft erst abends, wenn ich auf meine Tablettenbox schaue.“ Fenkl sagt, dass die regelmäßige Einnahme entscheidend ist, um die Viruslast klein zu halten. Auch Durchfall kann dazu führen, dass die Wirkstoffkombination im Körper sinkt und die Erreger trotz pünktlicher Einnahme die Oberhand gewinnen. Fenkl hat sich dadurch bereits Resistenzen eingefangen.
Auf der Suche nach Hilfestellung bei der Medikamentenwahl
Der Anstoß zur Entwicklung der Geno2Pheno-Software kam Ende der 1990-er Jahre von den Forschern der Universität Köln. „Wir hatten erkannt, dass mit Einführung der Kombinationstherapie der Bedarf für ein Werkzeug gewachsen war, das Hilfestellung bei der Medikamentenwahl gibt“, erinnert sich Rolf Kaiser, Virologe an der Kölner Uni. Die Forscher nahmen Kontakt zu den Bioinformatikern Niko Beerenwinkel und Joachim Selbig aus der Arbeitsgruppe von Thomas Lengauer auf, die sich damals noch am GMD-Forschungszentrum in Sankt Augustin befand und später zum Saarbrücker Max-Planck-Institut wechselte.
Beerenwinkel, der die Ergebnisse der Kölner und auch der Kooperationspartner aus Erlangen, die Arevir-Daten, in eine statistische Form goss, wurde für seine Entwicklung und die weiterführende Arbeit zum Thema HIV-Resistenz mit der Otto-Hahn-Medaille geehrt, dem wichtigsten Preis für Nachwuchswissenschaftler der Max-Planck-Gesellschaft. Inzwischen ist Beerenwinkel von Saarbrücken an die University of California in Berkeley gewechselt.
Stand: 07.04.2006