Den Medizinern helfen seit geraumer Zeit Forscher, die auf den ersten Blick mit der Virologie und den biologischen Vorgängen im Körper des Menschen so gar nichts gemein haben – Mathematiker vom Max- Planck-Institut für Informatik in Saarbrücken. Seit Ende der 1990er- Jahre befassen sich die Wissenschaftler mit der Anpassungsfähigkeit der HI-Viren. Im Blick haben sie jene Mutationen im genetischen Code der Viren, die zur Resistenz der Erreger führen.
Mithilfe mathematischer Verfahren bringen die Experten ihren Computern bei vorherzusagen, gegen welche Medikamente die Viren im Blut des Patienten mit hoher Wahrscheinlichkeit resistent sind – und welche Medikamente möglicherweise noch anschlagen. Inzwischen können die digitalen Diagnosewerkzeuge sogar abschätzen, wie schnell sich bei der einen oder anderen HAART-Kombination bei verschiedenen Patienten bestimmte Mutationen durchsetzen werden.
Letzte Rettung Geno2Pheno?
Bereits seit vier Jahren brummt auf den Servern in dem weißen Hochhaus am Rande der saarländischen Hauptstadt eine Rechenmaschine. Sie hilft Medizinern bei der Wahl der geeigneten Therapie. Geno2Pheno heißt der mathematische „Assistenzarzt“. Unter der Adresse www.geno2pheno.org können Mediziner aus der ganzen Welt darauf zugreifen. Entstanden ist das Regelwerk in Zusammenarbeit mit Virologen der Universität Köln und des Deutschen Referenzzentrums für Retroviren in Erlangen. Während die Saarbrücker für die Programmierung der Software zuständig waren, lieferten die Kölner und Erlanger anonymisierte Patientendaten aus der Arevir-Datenbank, die mit Unterstützung der Deutschen Forschungsgemeinschaft entstanden ist. Damit wurden die Computer gefüttert.
„Wir benutzen Methoden des statistischen Lernens, um die Daten auszuwerten“, sagt Tobias Sing, wissenschaftlicher Mitarbeiter am Max- Planck-Institut, „der Computer wird zunächst mit einem großen Datensatz trainiert und kann anschließend bei einer Abfrage die individuelle Patienteninformation entsprechend zuordnen.“ Doch wie bringt man einem Computer bei, die Krankheit zu verstehen? Indem man ihn zunächst mit genotypischen und phänotypischen Daten füttert. Die gewinnen Virologen aus dem Blut HIV-Infizierter.
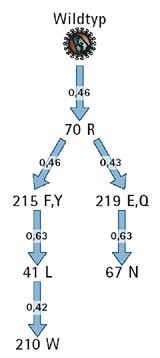
Bei der genotypischen Information handelt es sich um eine Analyse des genetischen Codes der Viren. Bekanntlich bestimmt die Abfolge der DNA-Bausteine, die Nukleinsäuresequenz, in welcher Reihenfolge sich Aminosäuren zu Eiweißen (Proteinen) zusammensetzen. Mutationen in der Nukleinsäuresequenz können einen Austausch von Aminosäuren im Protein bewirken. Mit Sequenziermaschinen, wie sie etwa die Kölner Experten betreiben, lässt sich die Abfolge der Nukleinsäuren und damit der Aminosäurebausteine bestimmen – und folglich auch, welche Mutationen vorliegen.
Auf der Suche nach den Virenvarianten
Bevor Ärzte eine neue HAART beginnen, wird bereits seit längerer Zeit genotypisch genau bestimmt, welche Virenvarianten der Patient in sich trägt. Zwar gibt es einen HIV-Wildtyp – die klassische Variante –, der in vielen Fällen nach wie vor den größten Teil der Virenlast im Körper ausmacht. Hinzu kommen aber verschiedene Virenmutanten in unterschiedlichen Anteilen. Mit der Sequenzierung lässt sich relativ zuverlässig bestimmen, welche Virenstämme vorliegen.
Bei der phänotypischen Untersuchung hingegen betrachtet man im Experiment, wie sich die genetische Information des Virus auf seine entscheidende Eigenschaft, die Resistenz (den Phänotyp) auswirkt. Ein Laborant mischt dazu im Reagenzglas die aus dem Blut gewonnenen Viren des Patienten mit einem HIV-Medikament. So bestimmt man, wie viel Wirkstoff nötig ist, um das Wachstum des Virus zu drosseln. Im Vergleich mit einem bekannten Viren-Referenzstamm zeigt sich dann, wie viel mehr Wirkstoff man braucht, um die Vermehrung der resistenten Mutante lahm zu legen. Auf diese Weise ergibt sich ein Maß für die Stärke der Resistenz: der Resistenz-Faktor (RF).
Stand: 07.04.2006