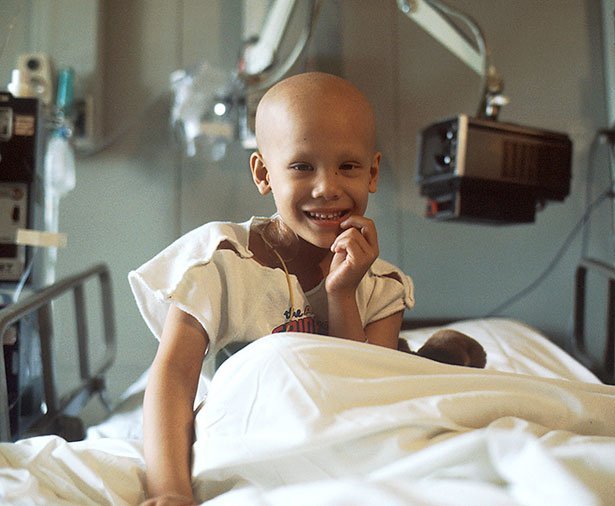
Die große Herausforderung besteht darin, medizinisches Datenmaterial in verwertbare Informationen zu transformieren. Wie ordnet und organisiert man beispielsweise Genom- oder Proteom-Daten?

„Handbuch“ ohne Inhaltsverzeichnis
„Als man im Jahr 2001 die Sequenzierung des menschlichen Genoms abgeschlossen hatte, freuten sich alle und glaubten, nun das Handbuch des Lebens zu kennen“, sagt Burkhard Rost, Professor für Bioinformatik an der TUM. „Aber wie bei allen Handbüchern ist es auch hier so, dass man das nicht findet, was man eigentlich braucht, und wenn man es findet, versteht man es nicht. Wir haben bei der Deutung der genetischen Sequenzen heute erst ein kleines Fenster in der Kathedrale des Wissens aufgestoßen.“
Lange herrschte zum Beispiel die Meinung, dass zwischen den Erbanlagen gesunder Menschen kein großer Unterschied besteht. Das Sequenzieren eines einzigen Genoms würde also ausreichen, um so gut wie alle Varianten zu erfassen. „Heute wissen wir, dass das falsch ist“, sagt Rost. „Betrachten wir einmal die Proteine, also die Eiweißstoffe in unserem Körper. Wir besitzen etwa 20.000 Varianten, und etwa ein Viertel davon unterscheidet sich bei zwei Personen ganz essentiell.“
Welche Therapie wirkt?
Solche Unterschiede können sich zum Beispiel darauf auswirken, wie und ob eine Chemotherapie wirkt. Manche Tumore sind gegen bestimmte Mittel unempfindlich, oder der Wirkstoff wird von den Krebszellen unschädlich gemacht. „Bei den meisten Massentumoren wie Brust-, Lungen-, Magen- und Darmkrebs spricht nur rund ein Fünftel der Patienten auf die konventionellen Chemotherapien gut an“, erklärt Bernhard Wolf, Professor für Medizinische Elektronik an der TU München.
„Um die Patienten nicht unnötig zu belasten, den Therapieerfolg zu steigern und damit langfristig auch Kosten zu senken, ist es deswegen notwendig, die Therapie zu personalisieren“, so der Forscher. Weil es bisher noch nicht gelungen ist, die Sensitivität der Patienten durch genetische Marker zuverlässig zu bestimmen, hat sein Team Sensoren entwickelt, die im Vorhinein messen, wie stark Zellen, die dem Tumor des Kranken entnommen wurden, auf verschiedene Chemotherapeutika reagieren. Entsprechend dieser Wirkung kann der Arzt dann denjenigen Wirkstoff anwenden, der individuell am besten geeignet ist.
Das neue System ist bereits einsatzbereit und wird derzeit in einer vorklinischen Studie in einer Kooperation mit der Asklepios Klinik in Hamburg-Barmbek getestet.
TU München
Stand: 05.01.2018