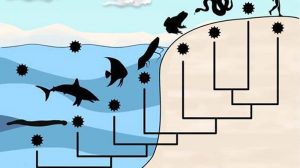
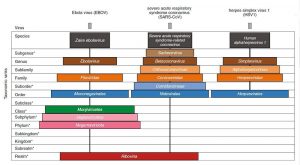
Es gibt auf unserem Planeten weit mehr Viren als lebende Zellen – und nahezu täglich werden neue entdeckt. Doch erst jetzt haben Forscher ein Schema entwickelt, um die virale Vielfalt zu kategorisieren und einzuordnen. Diese neue Taxonomie der Virosphäre ermöglicht es erstmals, übergeordnete Beziehungen verschiedener Virenarten sichtbar zu machen – auch das neue Coronavirus SARS-CoV-2 hat dort schon seinen Platz.
Viren sind überall und in fast allem: Es gibt auf unserem Planeten kaum ein Lebewesen, das nicht von Viren besiedelt wird. Trotzdem liegt das wahre Ausmaß der viralen Vielfalt noch im Dunkeln. Forscher schätzen, dass man jedem Stern im Kosmos 100 Viren zuordnen könnte – bekannt ist von dieser enormen Menge – einer Zahl mit 30 Stellen – aber bisher erst ein winziger Bruchteil. Umso wichtiger wäre es, wenigstens die bekannten Viren in einem einheitlichen Schema oder Stammbaum zu erfassen.
Warum ein Stammbaum so schwierig ist
Doch bisher gibt es eine solchen Virenstammbaum nicht. Zwar bekommen Viren Artnamen und werden auch Gattungen und Familien zugeordnet. So gehört SARS-CoV-2 zur Gattung der Betacoronaviren und zur Familie der Coronaviren. Aber oberhalb der Familien-Ebene fehlte bislang eine einheitliche Struktur. Ob und wie die Coronaviren beispielsweise mit den Ebolaviren zusammenhängen, ist daher unklar.
Einer der Gründe dafür: Die Evolution der Viren verläuft nicht linear. Anders als zelluläre Lebewesen geben sie ihr Erbgut nicht nur über Vermehrung weiter, sondern tauschen Gene untereinander und mit ihren Wirten aus. So tragen auch wir Menschen von Viren geerbte DNA-Abschnitte im Erbgut. Umgekehrt finden sich bei Viren Sequenzen, die von Bakterien, Tieren oder Pflanzen stammen. Das macht es fast unmöglich, Verwandtschaften anhand von Genvergleichen zu rekonstruieren. Zudem mutieren Viren erheblich schneller als alle zellulären Organismen, was die Einordnung zusätzlich erschwert.