Auch heute schon gibt es Verfahren zur automatisierten Analyse von DNS. Im Zentrum dieser Methodik steht die „erotische“ Anziehung zwischen komplementären DNS-Strängen. Im antiken Griechenland bezeichnete der Komödiendichter Aristophanes „Eros“ als die Kraft, die Mann und Frau als getrennte Hälften, die einstmals verbunden waren, wieder zueinander finden lässt.
{1l}
Auch die Erbsubstanz ist eine Einheit aus zwei Teilen. Trennt man die in Form einer Doppelhelix vorliegende Erbsubstanz in zwei Einzelstränge auf, bleiben die Teile solange alleine, bis sie sich mit ihrer „fehlenden Hälfte“ wieder zu einem Ganzen zusammenschließen können. Diese Eigenschaft machen sich Biotechnologen zu nutze. Auf DNS-Chips, auch „Biochips“ oder „Mikroarrays“ genannt, platzieren sie Tausende von kurzen DNS-Einzelsträngen mit bekannten Basensequenzen. Die fixierten Nukleotid-Einzelketten dienen gewissermaßen als Sonden, um in einem Gemisch von unzähligen DNS-Molekülen einer Patientenprobe die genetischen Gegenstücke zu identifizieren.
Sobald ein DNS-Fragment in Kontakt mit seinem fehlenden Partner gerät, verbinden sich die isolierten Einzelstränge wieder zu einer Doppelhelix. Alle Verfahren für die Hochdurchsatzanalyse von genetischem Material basieren auf dieser natürlichen Eigenschaft von DNS-Bausteinen, sich mit ihren fehlenden Partnerteilchen zu verbinden. Wie bei einem Puzzle fügen sich das Träger-Substrat und die gesuchte Ziel-DNS nach dem „Schlüssel-Schloss-Prinzip“ ineinander. Die Ansätze unterscheiden sich lediglich in der Art, wie dieses „Andocken’ detektiert wird.
Farbmarkierung verrät Gendefekt
Eine weit verbreiterte Technik ist die Markierung von DNS-Strängen mit fluoreszierenden Farbstoffmolekülen. Bindet ein markiertes Molekül an eine passende DNS-Sonde, entsteht nach Beleuchtung mit einem geeigneten Laser ein Raster aus Leuchtsignalen. Die fluoreszierenden Punkte signalisieren dabei die Anwesenheit eines bestimmten Gens oder eines bestimmten Gendefekts. Mit den einzelnen Leuchtsignalen lassen sich schließlich die Informationen über die Genaktivitäten in erkrankten oder gesunden Zellen erfassen.
Dieses Verfahren birgt allerdings gewisse Nachteile. So ist es aufwendig und kostenintensiv die DNS-Proben mit den entsprechenden fluoreszierenden Markern zu versehen. Darüber hinaus können die Farbstoffmoleküle den Nachweis von Genen beeinträchtigen, da die Leuchtsignale mitunter variieren. Eine eindeutige Quantifizierung der Anwesenheit eines Genes ist dann nicht möglich. In vielen Fällen, insbesondere in der Krebsforschung, ist es jedoch von entscheidender Bedeutung mengenmäßige Aussagen über die Anwesenheit von Genen oder Gendefekten machen zu können.
Einfacherer Nachweis ohne aufwändiges Färben
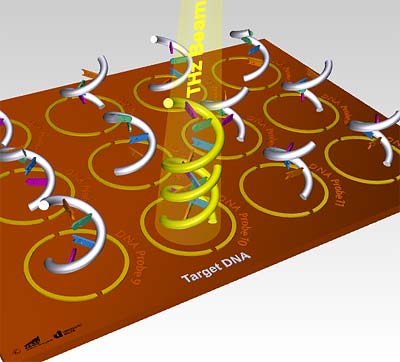
Für die Konstruktion eines leistungsfähigen DNS-Scanners ist das farbbasierte Nachweisverfahren problematisch. In der Summe erweist sich diese Detektionstechnik als zu arbeitsintensiv, zu kostenaufwendig und in einigen Applikationen als zu ungenau. In Vorexperimenten fand das Team um Haring Bolívar heraus, dass man mit elektromagnetischen Wellen von einigen Billionen Hertz – den THz-Strahlen – ohne Umweg herausfinden kann, ob zwei komplementäre DNS-Stränge gekoppelt vorliegen oder nicht.
Die Terahertz-Wellen (ein THz entspricht 10 hoch 12 Hz) regen in den DNS-Molekülen eine Reihe von charakteristischen Schwingungen an, die nur in gebundenen Gensequenzen möglich sind. Über die THz-Resonanzen, die als THz-Absorption oder THz-Refraktion messbar sind, kann der Bindungszustand zwischen bekannten und unbekannten DNS-Sequenzen direkt bestimmt werden. Damit wird es möglich THz Biochips aufzubauen, die ohne jegliche Markierung Gensequenzen direkt quantitativ detektieren und identifizieren können.
Stand: 04.01.2008