Allerdings ist es angesichts der Fülle von Medikamenten und Mutationen schwer, aus diesen Tabellen die geeigneten Arzneien für eine neue Therapie abzuleiten. Das Decision-Tree-Verfahren automatisiert diesen Entscheidungsprozess. Wie bei der Support-Vektor-Maschine (SVM) auch geht es hier darum, die Virensequenz aus einer Blutprobe des Patienten einer von zwei Klassen zuzuordnen – „resistent“ und „nicht resistent/suszeptibel“.
In der Trainingsphase füttern die Forscher den Algorithmus wiederum mit den Aminosäuresequenzen aus den Viren der 900 Referenzpatienten. In diesem Fall ordnen sie den Sequenzen jedoch keine genauen Resistenzfaktoren, sondern lediglich die Information „resistent“ oder „suszeptibel“ zu. Der Computer setzt dann alle 900 Sequenzen zueinander in Beziehung und zieht selbstständig logische Verknüpfungen zwischen den verschiedenen Mutationen.
Computer ermittelt Resistenzrisiko
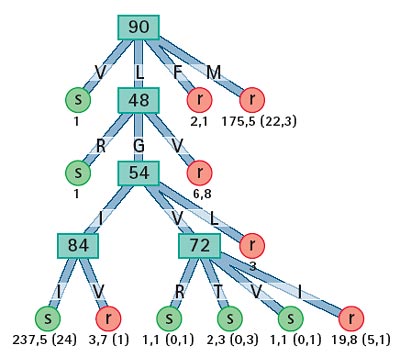
Füttert man den Decision-Tree-Algorithmus dann mit einer Patientensequenz, gleicht der Computer die Daten mit dem erlernten Wissen ab. Er weiß, bei welcher Kombination von Mutationen Resistenzen gegen ein Medikament auftreten. So liefert er einen Entscheidungsbaum, in dem man wie in einem Flussdiagramm von Schritt zu Schritt nachvollziehen kann, bei welcher Abfolge von Mutationen eine Resistenz vorliegt. Etwa so: „Befindet sich an Position 90 die Aminosäure Valin, ist das Virus nicht gegen das Medikament Saquinavir resistent. Befindet sich an Position 90 die Aminosäure Valin und an Position 48 ebenfalls die Aminosäure Valin, dann ist das Virus resistent.“
Je mehr Stationen der Decision Tree hat, desto komplexer wird das Beziehungsgeflecht. Desto deutlicher wird auch, wie schwer es ist, die Zusammenhänge allein mit Menschenverstand entwirren zu wollen. In Geno2Pheno setzen sich die SVM und der Decision-Tree-Algorithmus gleichzeitig in Bewegung, wenn man eine Sequenz eintippt. Die Ergebnisse zeigt der Computer dann in einer gemeinsamen Tabelle an.