Wissenschaftler haben nach 20 Jahren Suche nun herausgefunden, wie ein für Tomaten- und Paprika-Pflanzen spezifischer Krankheitserreger funktioniert. Wie sie in der Fachzeitschrift „Science“ berichten, ist der Mechanismus ungewöhnlich, denn das Bakterium nutzt Proteine um das Erbgut der befallenen Pflanzenzelle umprogrammieren.
Fataler Proteincocktail
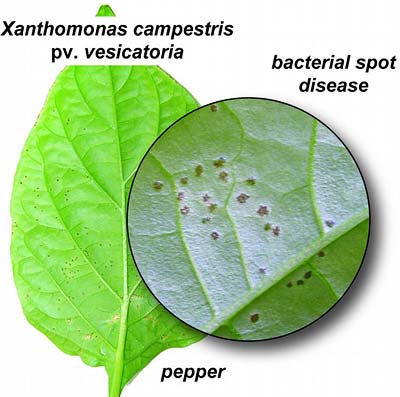
Bakterien, die Pflanzen befallen, injizieren einen Cocktail bakterieller Proteine über eine nadelartige Struktur in Pflanzenzellen hinein. Der wenig bekömmliche Cocktail bewirkt, dass die befallenen Pflanzen schneller altern und weniger Früchte tragen. Die entscheidende Rolle bei der Tomaten- und Paprikakrankheit spielt ein Protein namens AvrBs3.
„AvrBs3 ist ein Protein dieses Cocktails, das in Paprika- und Tomaten-Pflanzen Schäden anrichtet. Diese Cocktail-Komponente führt zu charakteristischen Pusteln auf den Blättern der Paprika-Pflanze“, erläutert Ulla Bonas. Zwar ist die Krankheit in unseren Breiten kein großes Problem, da die Winterfröste den Erreger abtöten. Aber in Regionen mit warmen und feuchtem Klima, zum Beispiel Florida und Israel, führt der Krankheitserreger zu immensen Verlusten. Das Gen, das dieses Protein kodiert, ist schon vor rund 20 Jahren isoliert worden, doch bisher war es nicht gelungen, seine genaue Funktion zu entschlüsseln.
Protein programmiert Pflanzengene um
Ulla Bonas und ihre Kollegen am Institut für Biologie der Universität Halle haben nun herausgefunden, wie das AvrBs3-Protein wirkt. „Es geht direkt in den Kern der Pflanzenzelle und weist als bakterielles Protein Eigenschaften auf wie ein Protein aus einem höheren Organismus. Aufgrund seiner Struktur ist das bakterielle Protein in der Lage, die Genregulation seines pflanzlichen Wirtes zu verändern. Es wirkt dabei im Zellkern, der Schaltzentrale der Pflanzenzelle, und nutzt die Maschinerie der Wirtspflanze zum eigenen Vorteil aus.“
Die Umprogrammierung bedingt, dass plötzlich Proteine in hoher Zahl produziert werden, die normalerweise nur auf niedrigem Niveau hergestellt werden. „Das ist, als wenn man die Produktion eines bestimmten Teiles in einer Fabrik hochfährt. Dieses Hochfahren haben wir nun beobachten können“, so die Hauptautorin der Studie.
Die meisten bakteriellen Proteine, die ihren Wirt manipulieren, greifen auf der Protein-Ebene an. „Dieses bakterielle Protein dreht jedoch an Schaltern, die im Kern zu finden sind, auf der Ebene der DNA, des Erbguts. Deshalb sind die Erkenntnisse wirklich neu und so spannend“, ergänzt Dr. Thomas Lahaye.
Resistenz durch „Opferzellen“
Es gbt allerdings auch Pflanzen, die gegen das Bakterien-protein resistenz sind: „Wir konnten zeigen, dass die Pflanze dann genau denselben Mechanismus nutzt wie in der nicht resistenten Pflanze“, erklärt Lahaye. „Das bakterielle Protein schaltet ein Gen ein, dass für einen lokalen Zelltod sorgt. Die Zellen, die mit dem Erreger sterben, opfern sich sozusagen. Somit wird die bakterielle Vermehrung gestoppt.“.
Bei den bisher bekannten Resistenzmechanismen der Pflanze ermöglichen pflanzliche Proteine die Erkennung des Krankheitserregers. „Hier erfolgt die Erkennung des Krankheitserregers über die DNA. Das Protein, das für den Zelltod sorgt, ist zunächst nicht vorhanden, es muss erst produziert werden. Es wird produziert, und es hat eine völlig neue Struktur – das ist das bahnbrechend Neue an unseren Ergebnissen.“
Anwendung auch bei anderen Pflanzenarten wahrscheinlich
Die Pflanzengenetiker betreiben zwar Grundlagenforschung, sehen aber gute Chancen für eine Anwendung ihrer neuesten Erkenntnisse. „Damit man so etwas anwenden kann, muss man die Mechanismen verstehen“, führt Lahaye aus. „Langfristig sind unsere Erkenntnisse für die Landwirtschaft von hoher Bedeutung, und es sind auch schon einige Firmen interessiert an dem, was wir machen. Zudem können viele Kollegen auf unseren Arbeiten aufbauen, zum Beispiel diejenigen, die mit Reis arbeiten.“
Es gebe viele Bakterien, die weltweit auf Reisfeldern große Schäden verursachen. Diese Bakterien enthalten Proteine, die AvrBs3 sehr ähnlich sind. „Was wir herausgefunden haben, ist nicht spezifisch für eine Pathogen-Familie.
Da der Mechanismus vermutlich auf breiter Ebene Gültigkeit hat, ist er so spannend und bedeutend.“
(Universität Halle-Wittenberg, 26.10.2007 – NPO)