Als der Mensch sich vor etwa 60.000 Jahren aus Afrika aufmachte, um die Welt zu besiedeln, war er nicht allein: Das Bakterium Helicobacter pylori, das heute bei vielen Menschen Magenschleimhautentzündungen auslöst, begleitete ihn. Und zusammen verbreiteten sich Mensch und Bakterium dann über die ganze Welt. Zu diesem Schluss kommt ein internationales Wissenschaftlerteam jetzt in „Nature“. Die Forscher stellten zudem fest, dass sich im Erbgut der Bakterienpopulationen genauso Unterschiede entwickelten wie in den verschiedenen menschlichen Völkern. Daraus gewinnen die Forscher auch neue Erkenntnisse, welche Wege die Völker auf der Erde einschlugen.
Mehr als die Hälfte aller Menschen sind mit Helicobacter pylori infiziert, einem Bakterium, das Magengeschwüre verursachen kann. Und wie die Menschen unterteilen sich auch die Bakterien in viele regionale Populationen. Ein Wissenschaftlerteam um Mark Achtman vom Max-Planck-Institut für Infektionsbiologie, François Balloux von der Universität Cambridge und Sebastian Suerbaum von der Medizinischen Hochschule Hannover fand nun Hinweise für eine parallele Evolution von Menschen und H. pylori.
Mit Hilfe mathematischer Simulationen zeigten die Forscher, dass H. pylori zum gleichen Zeitpunkt wie der Mensch aus Ostafrika ausgewandert sein muss – vor etwa 60.000 Jahren. Die erstaunliche Übereinstimmung entdeckten die Wissenschaftler, indem sie jeweils die Nukleotidsequenzmuster der DNA von menschlichen und H. pylori-Populationen verglichen.
Mutationsunterschiede als Messinstrument
Um die einzelnen Populationen zu charakterisieren, nutzten die Forscher das Prinzip der Isolation durch Abstand (isolation by distance) . Demnach hängt die genetische Distanz zwischen zwei Populationen linear mit der Länge der Wanderwege zusammen, die sie seit ihrer Trennung zurückgelegt haben. "Das ist eigentlich ganz logisch", erklärt Dr. Mark Achtman, "denn in der Zeit, die vergeht, seit eine Population ihr Ursprungsgebiet verlassen hat, nimmt die Zahl der Mutationen im Erbgut immer weiter zu."
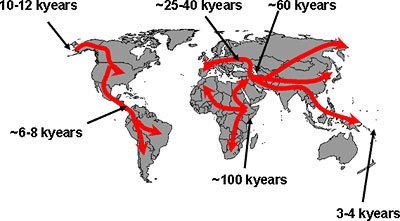
Doch während sich der Mensch über die Erde ausbreitete, mussten seine Populationen immer wieder sogenannte genetische Flaschenhälse passieren: Wenn eine Population schrumpft, wird auch der Gen-Pool kleiner. Und diese Einbußen an genetischer Vielfalt bleiben erhalten, wenn sich die Population wieder vermehrt. Da die Populationen von Homo sapiens auf ihrem Weg um die Welt gewöhnlich durch mehrere genetische Flaschenhälse hindurch mussten, nahm ihre genetische Vielfalt umso mehr ab, je weiter sie sich von ihrem Ursprung in Ostafrika entfernten.
Erstaunliche Parallelen
Die Wissenschaftler fanden nun ganz ähnliche Spuren dieser zurückliegenden Völkerwanderungen im Erbgut von H. pylori. Die genetische Diversität der Bakterien ist allerdings größer als die des Menschen. Das eröffnet den Forschern die Möglichkeit, aus H. pylori-Daten auch auf die Wanderungsbewegungen des Menschen zu schließen. "Die Parallelen zwischen der Ausbreitung des Menschen und H. pylori sind wirklich erstaunlich", so Achtman: "Wir könnten mit Hilfe dieses Bakteriums zusätzliche Informationen über noch kontrovers diskutierte Themen der menschlichen Geschichte bekommen, wenn wir H. pylori gemeinsam mit menschlichen Daten analysieren."
Beispielsweise breiteten sich die Populationen von H. pylori nach der Auswanderung aus Ostafrika örtlich begrenzt in Südafrika, Westafrika, Nordostafrika, Indien und Ostasien aus. Und die Gene der in Europa isolierten Bakterien zeigen zum Beispiel Einflüsse aus Zentralasien – ein Hinweis auf menschliche Einwanderer, die aus Asien nach Europa kamen.
(MPG, 09.02.2007 – NPO)