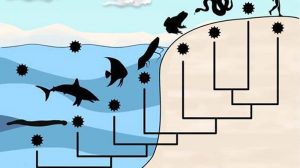
Nicht nur Pandemie-Erreger: Die Vielfalt der RNA-Viren ist weit größer als bislang angenommen, wie der Nachweis von tausenden zuvor unbekannten Viren im Ozean enthüllt. Durch sie bekommt der Stammbaum der RNA-Viren gleich fünf neue Stämme und elf Klassen hinzu – damit verdoppelt sich die Zahl der RNA-Virenstämme. Ein neuer Zweig dieser Viren könnte sich zudem als entscheidendes Bindeglied der Virenevolution erweisen, wie die Forschenden in „Science“ berichten.
Viren sind die ältesten und zahlreichsten Wesen auf unserem Planeten. Sie kommen nahezu überall vor und sind perfekt daran angepasst, sich in nahezu allen Lebensformen zu vermehren. Allein in unserem Körper tummeln sich Milliarden dieser Zellparasiten. Doch bisher ist erst ein winziger Bruchteil der gesamten Virenvielfalt bekannt. Versuche, die Virosphäre zu katalogisieren und ihren Stammbaum zu rekonstruieren, sind lückenhaft und unvollständig.
Fahndung nach viraler RNA
Dies gilt in besonderem Maße für die RNA-Viren – die Virengruppe, zu der das Coronavirus SARS-CoV-2 und die Erreger von Krankheiten wie Ebola, Masern, Mumps oder Hepatitis-D gehören. Sie gelten als besonders ursprünglich und könnten einigen Theorien zufolge sogar schon vor den ersten DNA-tragenden Zellen existiert haben. „RNA-Viren sind bedeutsam für unsere Welt, aber wir erforschen meist nur einen winzigen Teil von ihnen: die wenigen hundert Arten, die Menschen, Tiere und Pflanzen befallen“, erklärt Erstautor Matthew Sullivan von der Ohio State University.
Deshalb haben Sullivan und seine Kollegen eine großangelegte Virenfahndung in einem der bisher noch kaum auf RNA-Viren hin untersuchten Lebensräume durchgeführt: dem Ozean. Für ihre Studie analysierten sie die genetischen Daten von mehr als 35.000 Wasserproben aus allen Weltmeeren, die im Rahmen der Tara-Expedition gesammelt und sequenziert worden waren. Um die RNA-Viren zu erkennen, suchten die Forschenden nach einem Gen, RdRp, das nur bei diesen Viren vorkommt und daher eine Art Erkennungssignatur darstellt.