Das menschliche Protein hGBP1 (humanes Guanylat-bindendes Protein 1) ist an der Abwehr von Viren und anderen Krankheitserregern beteiligt. Wie das Eiweiß auf molekularer Ebene genau „arbeitet“, war jedoch bislang unbekannt. Diesen Mechanismus haben jetzt Wissenschaftler im Rahmen einer neuen Studie entschlüsselt.
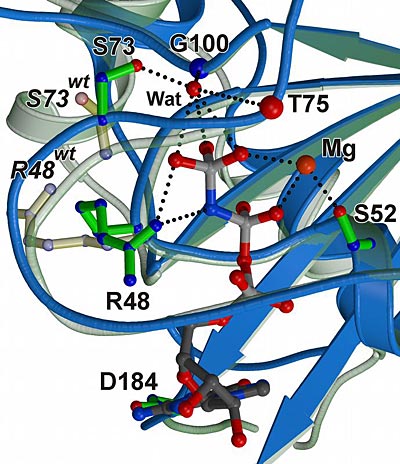
Danach besteht ein funktionelles Merkmal der Eiweißes hGBP1 in der katalytischen Spaltung von so genannten Cofaktor-Molekülen. Damit gehen die geordnete Zusammenlagerung und strukturelle Umwandlungen der Proteine einher, die für ihre biologische Wirkung von Bedeutung sind. Das von den Forschern erarbeitete Modell kann zum Verständnis der Funktionsweise einer Vielzahl ähnlicher Proteine dienen und Hinweise für die gezielte Behandlung verschiedener Krankheiten geben. Über ihre Ergebnisse berichten die Forscher in der aktuellen Ausgabe des Wissenschaftsmagazin NATURE.
Funktionsweise molekularer Maschine aufgeklärt
Das Enzym hGBP1 gehört zu einer Klasse von Proteinen, von denen einige eine wichtige Funktion bei der Abwehr von Viren haben, während andere für das Abschnüren von Membranbläschen im Innern der Zelle verantwortlich sind – dies dient der Aufnahme von Substanzen in die Zelle und der Regulation von Rezeptoren an der Zelloberfläche. Von hGBP1 ist eine antivirale Wirkung und ein Einfluss auf die Bildung von Blutgefäßen (Angiogenese) bekannt.
Charakteristisch für das Protein ist die Bindung und katalytische Spaltung eines Cofaktors, der einerseits die Struktur und damit die biologische Aktivität des jeweiligen Proteins reguliert. Zum anderen wird durch diesen Spaltungsvorgang bei einigen Proteinen aber auch die Energie für größere, strukturelle Änderungen und damit für die mechanische Arbeit dieser kleinen, molekularen Maschinen geliefert.