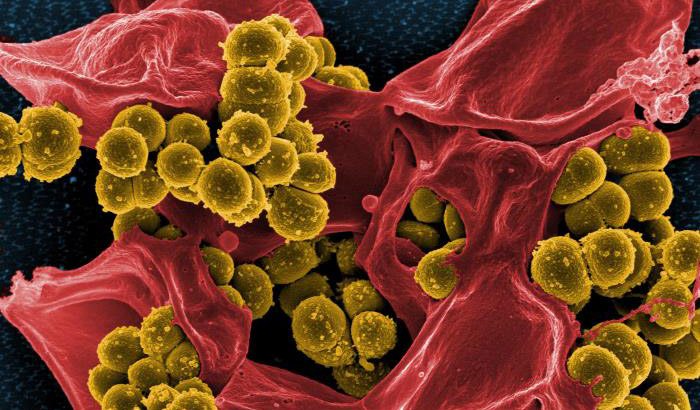
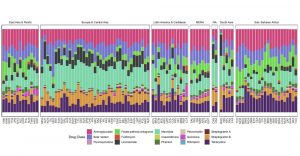
Schrankenlose Übertragung: Eine globale Kartierung von Antibiotika-Resistenzen enthüllt ein unerwartetes Muster der Ausbreitung. Demnach werden die Resistenzgene häufiger als erwartet auch zwischen sehr unterschiedlichen Bakterientypen weitergegeben. Ein besonderer Hotspot dieser gruppenübergreifenden Übertragung ist dabei das südliche Afrika, wie die Forschenden ermittelten. Gleichzeitig bilden die Resistenzen gegen verschiedene Antibiotika-Klassen trotzdem regionale Cluster.
Weltweit nimmt die Zahl resistenter und multiresistenter Bakterien immer weiter zu. Viele Erreger sind inzwischen selbst gegen Notfall-Antibiotika und neue Wirkstoffe immun. Als Folge starben allein im Jahr 2019 mehr Menschen an eigentlich heilbaren Infektionen als an HIV oder Malaria. Die von den Bakterien untereinander weitergegebenen Resistenzgene finden sich inzwischen überall im Boden, im Staub, in Gewässern und sogar in der Luft. Solange diese Gene in für uns harmlosen Bakterien vorkommen, schadet uns dies nicht, wohl aber, wenn sie in Krankheitserreger gelangen.
Fahndung im Abwasser
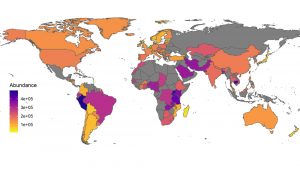
Wie weit verbreitet Resistenzgene inzwischen sind, haben nun Patrick Munk von der Technischen Universität Dänemarks und seine Kollegen untersucht. Dafür analysierten sie knapp 800 Abwasserproben aus 243 Städten in 101 Ländern, die zwischen 2016 und 2019 genommen worden waren. Ähnlich wie bei der Corona-Pandemie für das Coronavirus nutzten sie die Kanalisation in diesem Fall als aussagekräftiges Reservoir unseres bakteriellen Umfelds.
„Abwasser bietet uns eine gute, günstige Methode, um ganze Städte auf die in ihnen kursierenden Antibiotika-Resistenzgene hin zu beproben“, erklärt das Team. „Denn im Abwasser finden wir sowohl die Resistenzgene von Bakterien, die wir in uns tragen, als auch diejenigen, denen wir in Zukunft begegnen könnten.“ Die in den Proben gefundenen DNA-Sequenzen glichen die Forschenden mit einer Datenbank der mehr als 3.100 bisher bekannten Resistenzgene ab.