Unerkannte Gefahr: Das Coronavirus SARS-CoV-2 könnte schon in den 1960er Jahren entstanden sein, wie nun Erbgutanalysen enthüllen. Demnach spaltete sich der Pandemie-Erreger schon damals von seinem engsten Verwandten ab und kursierte dann jahrzehntelang in Fledermäusen, bevor er 2019 auf den Menschen übersprang. Wahrscheinlich gibt es noch weitere eng verwandte Fledermaus-Coronaviren mit dem Potenzial zum Artsprung, so die Forscher im Fachmagazin „Nature Microbiology“.
Wie SARS-CoV-2 entstand und woher es kommt, ist eine der großen Fragen der Corona-Pandemie. Genvergleiche legten schon früh nahe, dass sich die Vorfahren dieses Virus wahrscheinlich in Fledermäusen entwickelt haben. Als bislang ähnlichster Virenstamm gilt RaTG13, ein 2013 aus Java-Hufeisennasen in der chinesischen Provinz Yunnan isolierter Coronavirentyp. Weil diese Fledermausviren auch im Virologischen Institut in Wuhan erforscht werden, weckte dies zeitweilig den Verdacht, dass das SARS-CoV-2 in diesem Labor entstanden und von dort entwichen sein könnte.
Allerdings: Das für die Bindung an Menschenzellen wichtige Spike-Protein von SARS-CoV-2 hat eine noch größere Ähnlichkeit mit den Proteinen einiger bei Pangolinen vorkommenden Coronaviren. Einige Forscher vermuten deshalb, dass das Virus erst von Fledermäusen auf diese Schuppentiere übersprang und dann von dort aus den Artsprung zum Menschen machte.
Reger Genaustausch
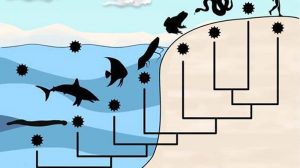
Ob das stimmt und wer die nächsten Verwandten von SARS-CoV-2 sind, haben nun Maciej Boni von der Pennsylvania State University und seine Kollegen näher untersucht. Dafür verglichen sie die RNA-Sequenzen von SARS-CoV-2, RaTG13 und weiterer Fledermaus-Coronaviren aus der Untergruppe der Sarbeco-Coronaviren. Die Forscher analysierten den Grad der Rekombination im Genom dieser Viren – Veränderungen, die auf einen gegenseitigen Austausch von Erbgut hindeuten.
Die Analysen enthüllten: SARS-CoV-2 und die gesamte Untergruppe dieser Coronaviren zeigen deutliche Hinweise auf einen regen Genaustausch untereinander und mit weiteren noch unbekannten Coronaviren. Das resultierende Mosaik aus RNA-Abschnitten unterschiedlicher Herkunft und die Zahl und Länge dieser „Mosaikteile“ ermöglichte es den Forschern, die Abstammung von SARS-CoV-2 und seinen Verwandten zu rekonstruieren.
RaTG13 ist der engste Verwandte
Das Ergebnis: SARS-CoV-2 und das Fledermausvirus RaTG13 sind am engsten miteinander verwandt – beide gehen auf einen direkten gemeinsamen Vorfahren zurück. Sowohl dieser Vorfahre als auch die beiden aktuellen Virentypen entwickelten sich wahrscheinlich in Hufeisennasen – einem Reservoir, in dem besonders viele eng verwandte Coronaviren vorkommen, wie Boni und seine Kollegen berichten.
Die beiden sehr ähnlichen, in Pangolinen gefundenen Coronaviren spalteten sich dagegen schon vor Entstehung der SARS-CoV-2/ RaTG13-Linie ab. Das aber bedeutet, dass SARS-CoV-2 sich wahrscheinlich nicht in Pangolinen zum Menschenvirus entwickelte, sondern in den Fledermäusen. „Pangoline könnten zwar als Zwischenwirte dienen, aber es gibt keine Belege dafür, dass die Fledermausviren diese Tiere benötigten, um den Artsprung zum Menschen zu schaffen“, so die Forscher.
SARS-CoV-2 ist schon vor Jahrzehnten entstanden
Doch wann ist SARS-CoV-2 entstanden? Antworten darauf lieferten RNA-Vergleiche kombiniert mit der wahrscheinlichen Rate der Mutationen und Rekombinationen der Coronaviren. „Nach unserem konservativsten Ansatz liegt der Zeitpunkt für die Aufspaltung von SARS-CoV-2 und RaTG13 im Jahr 1969“, berichten die Forscher. Mit zwei anderen Ansätzen kamen sie auf Zeiten um 1948 oder 1982. Auch der Erreger der SARS-Pandemie von 2002/2003 ist ihren Daten nach schon in den 1960er Jahren entstanden.
Das aber bedeutet, dass diese Coronaviren mehrere Jahrzehnte lang unter Fledermäusen kursierten, bevor sie zu Pandemie-Erregern wurden. Sie könnten demnach schon lange vor ihrem Artsprung das Potenzial zum Befallen des Menschen besessen haben – und sie sind möglicherweise nicht die einzigen: „Die lange Abspaltungszeit von 40 bis 70 Jahren legt nahe, dass es in Hufeisennasen noch weitere, bisher unerkannte Virenlinien gibt, die ein zoonotisches Potenzial besitzen“, konstatieren Boni und sein Team.
Zum Artsprung auf den Menschen kam es im Fall von SARS und SARS-CoV-2 aber offenbar erst, als günstige Bedingungen und ein enger Kontakt mit Menschen dies förderten. Wann und wo dies passierte, ist allerdings nach wie vor unklar.
Es könnte noch mehr potenzielle Pandemie-Erreger geben
Doch die Analysen enthüllten noch etwas: Die Strukturen, mit denen SARS-CoV-2 an den ACE2-Rezeptor auf menschlichen Zellen andockt, sind keine „Neuerfindung“ dieses Virus. Stattdessen sind große Teile seines Spike-Proteins auffallend wenig durch Rekombinationen verändert – und finden sich daher auch bei anderen eng verwandten Fledermaus-Coronaviren. Damit könnten diese Erreger auch bereits erste Anpassungen für den Befall menschlicher Zellen besitzen.
Nach Ansicht von Boni und seinen Kollegen sprechen diese Ergebnisse dafür, dass es noch weitere, potenziell pandemieträchtige Coronaviren in den Hufeisennasen geben könnte. „Die existierende Vielfalt und die dynamischen Prozesse der Rekombination zwischen den verschiedenen Fledermauslinien machen es jedoch schwer, Viren mit Epidemie-Potenzial schon vor ihrem Auftauchen zu identifizieren“, so die Forscher. Eine intensive Überwachung des Geschehens sei daher besonders wichtig. (Nature Microbiology, 2020; doi: 10.1038/s41564-020-0771-4)
Quelle: Nature Microbiology