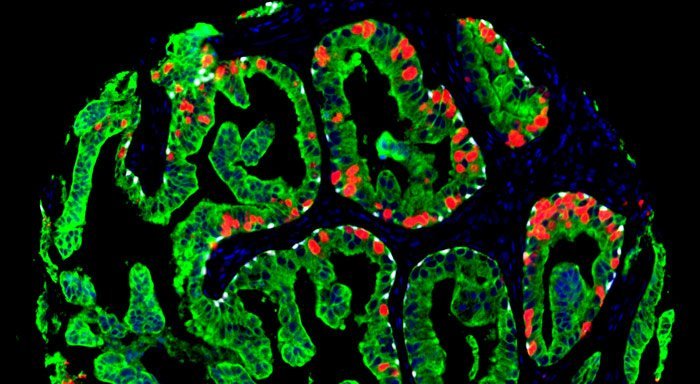
Abwarten oder sofort behandeln? Bei dieser Entscheidung könnte Prostatakrebs-Patienten und ihren Ärzten künftig ein Computermodell helfen. Dieses neue Prognose-Werkzeug analysiert charakteristische Mutationen und Genaktivitätsmuster im Krebsgewebe – und trifft auf Basis dieser Informationen Vorhersagen über den wahrscheinlichen Krankheitsverlauf. Auf diese Weise kann in Zukunft besser zwischen aggressiven und eher harmlosen Tumoren unterschieden werden, wie Forscher berichten.
Prostatakrebs ist eine der häufigsten Krebserkrankungen älterer Männer und die dritthäufigste Ursache für krebsbedingte Todesfälle. Allein in Deutschland wird diese Tumorform bei rund 60.000 Männern pro Jahr neu diagnostiziert. Viele der Betroffenen stehen dann vor einer schwierigen Entscheidung: Unterziehen sie sich sofort einer Therapie mit möglicherweise erheblichen Nebenwirkungen – oder entschließen sie sich, zunächst abzuwarten?
Spurensuche im Erbgut
Das Problem dabei: Bisher war es nur schwer möglich, zwischen eher harmlosen, langsam voranschreitenden und aggressiven Typen von Prostatakrebs zu differenzieren. Um Kriterien für diese Unterscheidung zu identifizieren und Patienten künftig besser beraten zu können, haben Wissenschaftler um Clarissa Gerhäuser vom Deutschen Krebsforschungszentrum in Heidelberg nun nach Hinweisen im Genom von Prostatatumoren gesucht.
Für ihre Studie analysierten sie die Sequenz und die chemischen Veränderungen der Erbinformation sowie die Genaktivität im Gewebe von fast 300 Krebspatienten. Durch den Vergleich von frühen und weiter fortgeschrittenen Tumoren konnten sie dabei beobachten, wie sich eine Prostatazelle auf dem Weg zur Entartung verändert – und welche Mutationen typisch für besonders aggressive Varianten sind.