„Um die gefährlichen von den harmlosen DNA-Ringen zu unterscheiden und ihre Evolution innerhalb des Tumors nachvollziehen zu können, muss man sich das Gewebe Zelle für Zelle anschauen“, erklärt Seniorautor Anton Henssen von der Charité. „Wir können dann auszählen, wie viele Zellen des Tumors einen spezifischen Ring beherbergen. Sind es wenige, ist der Ring nicht besonders relevant für das Krebswachstum. Sind es viele, verleiht er einer Tumorzelle offenbar einen Selektionsvorteil.“
Am Beispiel von Zellen des Neuroblastoms – eines vor allem bei Kindern auftretenden Hirntumors – haben González und ihre Kollegen die DNA-Ringe mit dieser Methode untersucht.
Enorme Vielfalt
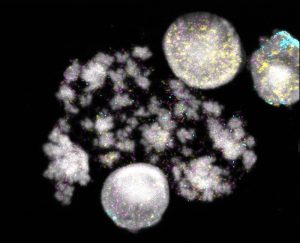
Es zeigte sich: In Bezug auf die extrachromosomalen Ringe ist keine Krebszelle wie die andere. Sie unterscheiden sich in Art, Größe und Anzahl der DNA-Ringe. Während in einer Tumorzelle nur knapp 100 solcher Ringe schwimmen, können es in der nächsten fast 2.000 sein, wie das Team feststellte. Große Unterschiede gab es auch bei der Größe der Ringe: Die kleinsten von ihnen enthielten nur rund 30.000 DNA-Basen, die längsten waren 1,2 Millionen DNA-Basen lang.
Nähere Analysen ergaben, dass auch der genetische Inhalt dieser extrachromosomalen Ringe sehr unterschiedlich ist: Die kleinen Ringe enthalten keine kompletten Gene, sondern meist nur regulatorische Elemente – DNA-Abschnitte, die das Ablesen bestimmter Gene beeinflussen. „Die kleinen Ringe haben wir nur vereinzelt entdeckt, sie haben für die Krebszellen wohl keine große Relevanz“, sagt González. Denn sonst würden sie bei den Zellteilungen konsequent weitergegeben und vermehrt.
Aus dem Chromosom gelöste Onkogene
Anders ist dies mit den großen DNA-Ringen: „Sie sind beladen mit Krebsgenen, die ursprünglich aus den Chromosomen der Zelle stammen“, erklärt González. „Diese Krebsgene haben sich sozusagen selbständig gemacht. Durch die Ringform umgehen sie die klassischen Gesetze der Genetik – und werden ein Stück weit autonom.“ Die Analysen zeigten, dass diese großen DNA-Ringe Onkogene und Genbruchstücke aus verschiedenen Chromosomen in sich tragen können.
„In unserer Studie haben wir die großen DNA-Ringe in vielen Neuroblastomzellen gefunden, sie treiben das Zellwachstum also offenbar an“, so González weiter. Offenbar ist es für die Krebszellen von Vorteil, wenn sie viele dieser zusätzlichen Onkogen-Kopien in sich tragen. „Welche Konsequenzen das hat, beginnen wir aber gerade erst zu verstehen.“
Optimierung bei jeder Zellteilung
Doch die DNA-Ringe bleiben selbst innerhalb einer Krebszelllinie nicht gleich. Stattdessen scheint es während des Tumorwachstums eine Art Evolution zu geben, wie das Team beobachtete: In den ersten Neuroblastomzellen löste sich zunächst nur das bekannte Krebsgen MYCN aus seinem Heimat-Chromosom heraus und bildete einen Ring. Dann verschmolzen zwei solcher Ringe zu einem größeren, der mehrere Teile seiner DNA abschnürte.
Im Laufe weiterer Zellteilungen entstanden so unterschiedliche Varianten dieser Onkogen-tragenden DNA-Ringe. „Erst der letzte Ring scheint einen Wachstumsvorteil mit sich gebracht zu haben, weil nur er in vielen Zellen des Neuroblastoms zu finden ist“, sagt Henssen. „Das zeigt, dass sich das Krebsgen durch diese Vorgänge nicht nur selbständig gemacht, sondern auch immer weiter ‚verbessert‘ hat.“ Ein solcher Einblick in die Evolution von DNA-Ringen innerhalb eines Tumors wäre ohne die neu entwickelte Methode nicht möglich gewesen.
Hoffnung auf bessere Diagnose und Therapie
Das Forschungsteam möchte die neu entwickelte Auslesetechnik nun nutzen, um die Entwicklungsschritte der DNA-Ringe bei weiteren Krebsarten zu rekonstruieren und die Auswirkungen zu untersuchen. Das könnte dabei helfen, die gefährlichen von den harmlosen DNA-Ringen zu unterscheiden. Mit der Erforschung dieser speziellen Form der Erbinformation in Tumorzellen verbinden Wissenschaftler weltweit die Hoffnung auf neue Therapieansätze gegen Krebs.
„Unsere Hoffnung ist, dass wir in Zukunft durch einen Blick auf die DNA-Ringe im individuellen Fall erkennen können, ob der Tumor besonders aggressiv ist oder nicht“, sagt Henssen. „Dann könnten wir die Therapie daran anpassen. Die Vorhersagekraft von spezifischen DNA-Ringen zu testen, ist deshalb unser nächstes Forschungsziel.“ (Nature Genetics, 2023; doi: 10.1038/s41588-023-01386-y)
Quelle: Charité – Universitätsmedizin Berlin
8. Mai 2023
- Nadja Podbregar