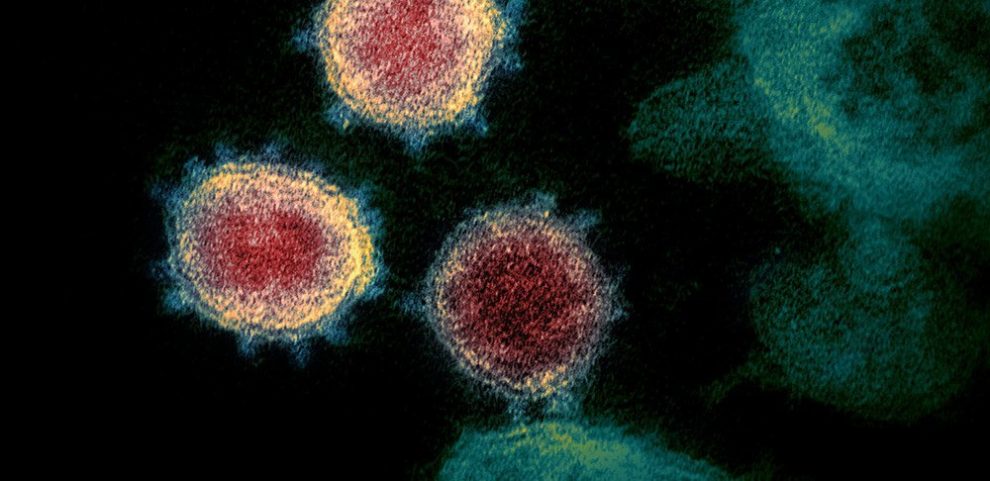
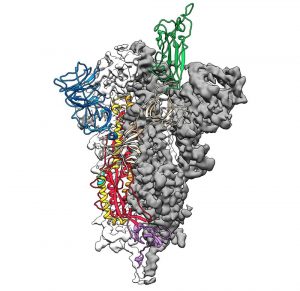
Ansatzstelle für Gegenmittel: Forscher haben erstmals die Struktur des Bindungsproteins entschlüsselt, mit dem das neue Coronavirus an menschliche Zellen andockt. Die atomgenaue 3D-Struktur sowie Experimente enthüllen, dass dieses Spike-Protein zwar dem der SARS und MERS-Erreger sehr ähnelt, aber anders reagiert. Dieses Wissen könnte nun die Entwicklung von Impfstoffen und antiviralen Mitteln gegen SARS-CoV-2 erleichtern.
Das neue Coronavirus SARS-CoV-2 hat nicht nur den Artsprung vom Tier zum Menschen geschafft, es kann auch relativ leicht von Mensch zu Mensch überspringen. Mediziner schätzen, dass das Virus in etwa so ansteckend ist wie die Grippe. Weil der Erreger zudem nicht immer Symptome verursacht und auch schon in der Inkubationszeit übertragbar ist, ist die Eindämmung der Epidemie erschwert. Forscher weltweit arbeiten daher mit Hochdruck daran, Impfstoffe und antivirale Mittel gegen das Virus zu finden.
Andock-Protein im Visier
Wichtiger Ansatzpunkt für solche Gegenmittel ist ein bestimmtes Oberflächenprotein des Coronavirus. Mit ihm dockt es an einen Rezeptor auf den menschlichen Zellen an und kann so in sie eindringen. „Das Spike-Glycoprotein der Coronaviren ist ein Schlüsselziel für die dringend benötigten Impfstoffe, therapeutischen Antikörper und Diagnostika“, erklären Daniel Wrapp von der University of Texas in Austin und seine Kollegen.
Damit das Andocken funktioniert, verändert das Coronavirus die Konformation seines Spike-Proteins. Es klappt auf und bringt sich so in eine für den Zellrezeptor passende Form. Das Problem jedoch: Diese Konformation ist instabil und lässt sich daher nicht ohne weiteres abbilden oder strukturell analysieren. Um dies zu umgehen, fügten die Forscher in die genetische Bauanleitung für dieses Virenprotein eine Mutation ein, die eine Aminosäure austauscht. Durch diese Änderung wird das Spike-Protein stabiler.