Verborgene Aktivität: In unserem Erbgut liegen unzählige Genabschnitte uralter Viren. Jetzt belegt eine Analyse, dass diese endogenen Retroviren aktiv abgelesen und transkribiert werden – und das in gesundem Gewebe unseres ganzen Körpers. Besonders hoch ist die Aktivität der genetischen Virenreste in unserem Gehirn und den Fortpflanzungsorganen. Was sie dort genau tun und welche Auswirkungen die Expression dieser uralten Viren-DNA hat, ist jedoch bisher unbekannt.
Das menschliche Erbgut enthält eine Reihe von Sequenzen, die dort eigentlich nichts zu suchen haben: Gene und Genfragmente von Viren. Diese humanen endogenen Retroviren (HERV) machen rund acht Prozent unserer DNA aus und stehen schon länger im Verdacht, unsere Gesundheit zu beeinflussen. So könnte die Reaktivierung dieser urzeitlichen Erreger Krebstumore verursachen und neurologische Krankheiten begünstigen. Umgekehrt scheinen einige HERVs auch vor Krebs zu schützen und das Muskelwachstum von Männern zu fördern.
Fahndung in 54 Geweben von 1.000 Menschen
Unklar war jedoch bisher, ob und wann die Virenreste in unserem Erbgut abgelesen werden – und in welchen Geweben. Wichtig ist dies, weil die endogenen Retroviren noch ganze Virengene enthalten können, aber auch DNA-Sequenzen, die die Aktivität benachbarter menschlicher Gene beeinflussen. Um dies zu klären, haben Aidan Burn von der Tufts University in Boston und seine Kollegen Proben von 1.000 Menschen auf die Aktivität von endogenen Retroviren aus der Gruppe HERV-K (HML-2) hin untersucht.
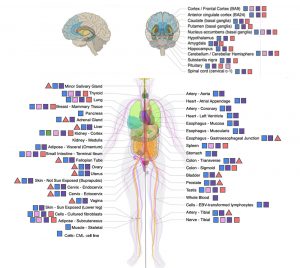
Diese Gruppe von Virenresten in unserem Genom stammt von urzeitlichen Erregern, deren älteste Vertreter vor rund 35 Millionen Jahren in unser Genom gelangten, die jüngsten wurden vor 200.000 Jahren in die menschliche DNA integriert. HML-2 finden sich überall in unserem Körper. Um der Aktivität dieser HERVs auf die Spur zu kommen, analysierten die Forscher 54 verschiedene Gewebearten aus unterschiedlichen Regionen des Körpers. Außerdem umfassten die Proben sowohl gesundes als auch krankes Gewebe.
Ableseprodukte endogener Retroviren in allen Geweben
Das überraschende Ergebnis: Die Virenreste der HML-2-Gruppe waren in allen Geweben und Körperteilen aktiv – egal, ob das Gewebe gesund oder krank war. Insgesamt detektierten die Forscher die RNA-Signaturen von 37 verschiedenen HML-2-Virenresten. Die RNA von fünf dieser endogenen Retroviren war in nahezu allen Gewebe nachweisbar, andere HML-2-Virenreste waren nur in bestimmten Geweben aktiv, wie das Team feststellte.
„Die Präsenz von HML-2-Transkripten und teilweise auch viralen Genen kann demnach als normaler Teil unseres Proteoms und Transkriptoms angesehen werden – auch in gesundem Gewebe“, konstatieren Burn und seine Kollegen. Welche dieser endogenen Retroviren wo aktiv sind und bei wem, hängt dabei offenbar vom Alter, vom Geschlecht und auch vom Virenrest ab.
Evolutionär ältere Virenreste sind aktiver
Dabei zeigte sich ein klarer Zusammenhang mit dem Alter der Virenreste: Diejenigen, die sich schon bei den gemeinsamen Vorfahren aller Menschenaffen und Menschen ins Erbgut eingeschleust haben, sind heute bei uns Menschen am aktivsten. Sie werden in fast allen Geweben abgelesen, wie die Analysen ergaben. Die erst bei den gemeinsamen Vorfahren von Mensch und Schimpansen oder sogar erst den Menschen akquirierten Retroviren sind hingegen nur in wenigen Geweben aktiv.
„Diese Daten sprechen dafür, dass HERVs unterschiedlichen Alters unterschiedlich stark vom menschlichen Genom kontrolliert werden“, erklären Burn und seine Kollegen. Ältere Virenreste hatten mehr Zeit, sich durch Mutation und Selektion an unsere DNA und Zellmaschinerie anzupassen. Sie könnten deshalb mehr Möglichkeiten haben, die Kontrollmechanismen zu unterlaufen und aktiv zu werden. Gleichzeitig könnten diese HERVs so lange erhalten geblieben sein, weil sie positive Auswirkungen auf unsere Zellen und Gewebe haben.
Höchste Expression im Gehirn und den Hoden
Unterschiede gibt es auch in Bezug auf die Gewebe: „Die HML-2-Expression variierte je nach Körperteil erheblich“, berichten Burn und seine Kollegen. Die mit Abstand höchste Aktivität der endogenen Retroviren fanden sie im Kleinhirn, der Hypophyse, den Hoden und der Schilddrüse. Auch andere Teile des Gehirns und Nervensystems zeigten eine relativ hohe Zahl aktiver HML-2-Virenreste. Kaum abgelesen wurden HERVs hingegen im Blut und in der Bauchspeicheldrüse.
„Diese Unterschiede demonstrieren, dass die Intensität der Expression für die HML-2-Gruppe gewebespezifisch ist – einige Gewebe und Organe begünstigen das Ablesen dieser Virenreste offenbar mehr als andere“, konstatieren die Forscher. Sie vermuten, dass die Virenreste in der Schilddrüse und Hypophyse durch die dort produzierten Hormone aktiviert werden.
Die hohe Aktivität in den Hoden könnte dagegen mit der Infektionsgeschichte dieser Viren zusammenhängen. Nach gängiger Theorie gelangten die meisten endogenen Retroviren und im Speziellen HML-2 in unser Erbgut, als das entsprechende Retrovirus die Keimzellen infizierte und sich in ihr Erbgut einbaute. Über die so infizierten Spermien und Eizellen vererbten sich diese Virenreste dann weiter an die nachkommenden Generationen. „Die prädominante Expression von jüngeren Proviren im reproduktiven Gewebe könnte dies widerspiegeln“, erklären die Forscher.
„Noch immer ein Rätsel“
Insgesamt unterstreichen diese Ergebnisse, dass die endogenen Retroviren in unserem Erbgut aktiver sind als lange angenommen. Diese Virenreste werden selbst in gesundem Gewebe abgelesen und könnten daher unzählige noch unerkannte Einflüsse auf unsere Gesundheit haben – sowohl positive wie auch negative. „Die Rolle der HERVs in der menschlichen Biologie ist noch immer ein Rätsel“, konstatieren Burn und sein Team.
„Es ist noch mehr Forschung nötig, um die Mechanismen ihrer Expression zu verstehen und auch die Unterschiede in der HERV-Aktivität zwischen gesundem und krankem Gewebe“, so die Forscher weiter. „Wichtig ist aber sich ins Gedächtnis zu rufen, dass HML-2-Transkripte im menschlichen Gewebe immer zu finden sind.“ Frühere Annahmen, nach denen aktive Virenreste sich vielleicht als Krankheitsmarker eigenen, müssen dies berücksichtigen. (PLoS Biology, 2022; doi: 10.1371/journal.pbio.3001826)
Quelle: PLOS