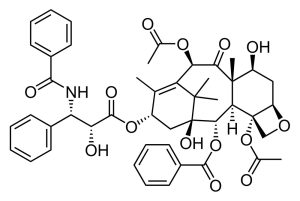
Biosynthese aufgeklärt: Forschende haben herausgefunden, wie Eiben das Krebsmedikament Paclitaxel produzieren. Dieses komplexe Molekül wird häufig eingesetzt, um bösartige Tumore zu behandeln, ist jedoch nur schwer künstlich herzustellen – und entsprechend teuer. Die jetzt gelungene Entschlüsselung des natürlichen Syntheseweges dieses Chemotherapeutikums in Pflanzen könnte nun die industrielle Produktion dieses Wirkstoffs beschleunigen.
In der Krebstherapie kommen regelmäßig giftige Chemikalien zum Einsatz, die Tumore abtöten. Leider sind diese Moleküle oft sehr komplex, schwer zu beschaffen und daher teuer. Eines dieser Chemotherapeutika ist das natürliche Pflanzenheilmittel Paclitaxel, das aus Eiben (Taxus brevifolia) gewonnen wird. Unter dem Namen Taxol ist es seit über 30 Jahren als Krebsmedikament zugelassen. Das Molekül hemmt die Zellteilung und tötet damit Tumorzellen ab. Allerdings werden für die Behandlung eines einzigen Menschen mit zwei Gramm Paclitaxel zehn Tonnen Nadeln der langsam wachsenden Eibe benötigt.
Chemische Synthese nach Vorbild der Natur
Paclitaxel künstlich im Labor herzustellen, ist zwar möglich, aber noch teurer als die Gewinnung aus Nadeln und Rinde. Denn aufgrund seiner komplizierten chemischen Struktur erfordert die Synthese dieses kreisförmigen Diterpenoids viele komplexe Schritte. Derzeit wird das Medikament deshalb entweder halbsynthetisch hergestellt – ausgehend von einer Zwischenstufe, die aus Eiben gewonnen wird – oder in pflanzlichen Zellkulturen. Beide Prozesse bleiben jedoch aufwendig und teuer. Forschende suchen daher seit Längerem nach anderen Produktionsmethoden für diesen Wirkstoff.
Ein typischer Ansatz in der Arzneimittelforschung ist es, den biologischen Syntheseweg der Pflanzen zu kopieren und dann im Labor in anderen Organismen effizienter nachzubauen. Dafür müssen die Forschenden jedoch alle Enzyme und ihren genetischen Code kennen, um den betreffenden Prozess zu kopieren. Bei Eiben war jedoch bislang nur in Teilen bekannt, mit welchen Enzymen und über welche Zwischenprodukte die Bäume Paclitaxel herstellen. Frühere Studien legten nahe, dass mindestens 20 verschiedene Enzyme beteiligt sind. Wie diese genau aussehen, war jedoch für einige Schlüsselreaktionen unklar.
Welche Enzyme verwendet die Eibe?
Eine Forschungsgruppe um Youjun Zhang vom Max-Planck-Institut (MPI) für molekulare Pflanzenphysiologie in Potsdam hat nun die noch fehlenden Schritte der Paclitaxel-Biosynthese untersucht und aufgeklärt. Dafür analysierten die Wissenschaftler die Funktion von mehreren zehntausend Genen in Eiben. In diesen Daten suchten sie dann gezielt nach Enzymen, die Eiben nur in sehr geringen Mengen herstellen – ähnlich, wie die zwölf bereits bekannten an der Biosynthese von Paclitaxel beteiligten Enzyme und Zwischenprodukte.
So schränkten die Wissenschaftler ihre Suche auf 37 potenziell an der Paclitaxel-Synthese beteiligten Enzyme ein. Mit weiteren chemischen Analysen und molekularbiologischen Werkzeugen untersuchten Zhang und sein Team dann diese Bausteine genauer. Sie entschlüsselten deren Funktion und fanden heraus, welche Enzyme in welcher Reihenfolge im Biosyntheseweg von Paclitaxel vorkommen.
Nachbau der Biosynthese in Pflanzen
Mithilfe dieser Daten gelang es Zhang und seine Kollegen erstmals, den gesamten Paclitaxel-Biosyntheseweg der Eibe zu entschlüsseln und nachzubauen. Dafür kopierten sie alle notwendigen Gene für die Eiben-Enzyme in die Modellpflanze Nicotiana benthamiana, einen in Australien vorkommenden Verwandten der Tabakpflanze. Die auf diese Weise umgerüsteten Pflanzen produzierten dann tatsächlich Paclitaxel, allerdings in noch geringeren Mengen als die Eibe, wie die Forschenden berichten.
Doch mit dem Wissen um dessen Biosyntheseweg könnte die Paclitaxel-Produktion nun beschleunigt werden. „Die Entdeckung des vollständigen Paclitaxel-Biosynthesewegs öffnet die Tür für die angewandte Forschung zur Optimierung der Produktion“, sagt Seniorautor Alisdair Fernie vom MPI für molekulare Pflanzenphysiologie.
Pflanzen-Enzyme funktionieren nicht in Bakterien
Um eine kostengünstige Produktion in großem Maßstab zu ermöglichen, wäre es jedoch nötig, den Prozess so anzupassen, dass er auch in Bakterien funktioniert. Die Forschenden versuchten daher auch, den Paclitaxel-Stoffwechselweg in Bakterien oder Hefen zu kopieren. Dabei stellten sie aber fest, dass einige der Enzyme in diesen Einzellern nicht funktionieren.
Das könnte mit der Lage der Enzyme in den Pflanzenzellen zu tun haben, vermuten Zhang und seine Kollegen. „In Pflanzen sind die meisten Enzyme, die an der Biosynthese von Paclitaxel beteiligt sind, an eine bestimmte Membran gebunden“, erklärt Zhang. „So liegen sie dicht beieinander und bilden wahrscheinlich eine Transportkette, in der jedes Enzym das Produkt des vorhergehenden übernimmt und es weiter modifiziert, bis am Ende das endgültige Paclitaxel freigesetzt wird.“ Da Bakterien andere Membranen haben als Pflanzen, könne es sein, dass die Enzyme dort nicht zueinander finden und daher nicht effektiv zusammenarbeiten können, vermuten die Forschenden.
Außerdem sei es möglich, dass die Modellpflanze Nicotiana benthamiana weitere hilfreiche, aber bislang unbekannte Begebenheiten für die Paclitaxel-Produktion bereitstelle, die in Bakterien fehlen. Ob sich dieses Problem umgehen lässt, müssen weitere Studien klären. „Bis dahin können die von uns geschaffenen Nicotiana-bethamiana-Linien zur Optimierung des Systems und zur Steigerung der Ausbeute bestehender Produktionslinien verwendet werden“, sagt Fernie. (Molecular Plant, 2023; doi: 10.1016/j.molp.2023.10.016)
Quelle: Max-Planck-Gesellschaft