Sechs Basen statt vier: Forscher haben ein Bakterium geschaffen, das zwei zusätzliche DNA-Basen in seinem genetischen Code trägt. Sein Gencode besteht damit aus sechs statt vier Buchstaben. Im Gegensatz zu Vorgänger-Exemplaren kann diese semisynthetische Mikrobe ihre künstlichen Basen nahezu unbegrenzt an ihre Nachkommen weitergeben und ist auch ohne die Gabe spezieller Zusatzmoleküle lebensfähig, wie die Forscher berichten.
Das Alphabet des Lebens besteht aus vier Buchstaben – und das schon seit Entstehen der ersten lebenden Zelle. Die Abfolge der Basen Guanin, Cytosin, Thymin und Adenin in der DNA kodiert die Baupläne für Proteine und unzählige Steuermoleküle unseres Zellstoffwechsels. Im Jahr 2014 jedoch gelang es Floyd Romesberg vom Scripps Research Institute in La Jolla und seinem Team, erstmals zwei synthetische DNA-Buchstaben in den Code des Darmkeims Escherichia coli einzuschleusen.
Doch diese ersten halbkünstlichen Mikroben waren nur bedingt lebensfähig: „Die Mikroben wuchsen schlecht, benötigten spezielle Bedingungen und schafften es selbst dann nicht, die unnatürliche Information längere Zeit zu behalten“, erklären Romesberg und seine Kollegen.
Optimierte Synthetik-Basen
Jetzt haben die Forscher diese Probleme mit einem neuen „Modell“ ihrer semisynthetischen Bakterien überwunden. „Wir haben nun einen semisynthetischen Organismus erschaffen, der autonomer ist als sein Vorgänger und der die zusätzliche Information fast genauso dauerhaft speichert wie die natürlichen DNA-Komponenten“, berichten die Wissenschaftler.
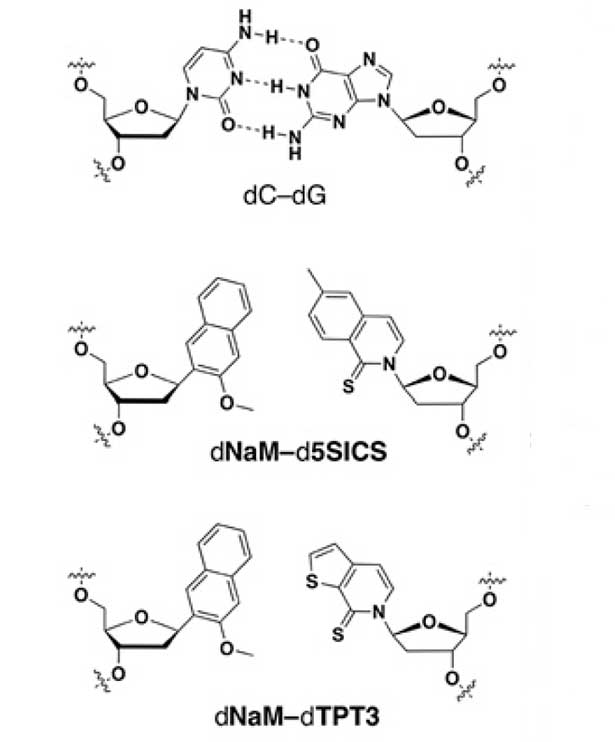
Für ihren neuen semisynthetischen Organismus schleusten die Forscher wieder zwei zusätzliche, künstliche DNA-Basen in das ringförmige Plasmid von Escherichia coli-Bakterien ein. Eine der Basen, dNaM, entsprach der des Vorgängermodells. Statt des früheren Gegenparts d5SICS verwendeten die Forscher nun jedoch das stabilere, ähnlich strukturierte Molekül dTPT3. „Dieses nichtnatürliche Basenpaar erlaubt es, Informationen mit einem aus sechs Buchstaben bestehenden genetischen Alphabet stabil zu speichern“, so Romesberg und seine Kollegen.
Vermehrung auch ohne Zusatzstoffe
Während der Vorgänger noch ein von außen zugegebenes Transportermolekül zur Vermehrung benötigte, ist dies nun nicht mehr der Fall. Die Forscher schleusten ein Gen in das Bakterienerbgut ein, das die Bauanleitung für das Transportermolekül enthält. Dadurch kann sich die semisynthetische Mikrobe nun ohne Zusatz dieses Moleküls vermehren und baut selbstständig Kopien der künstlichen Basen in die DNA seiner Nachkommen ein.
Und noch etwas erleichtert die Weitergabe der synthetischen Genbuchstaben: Die Mikrobe produziert nun ein Enzym, das alle Genkopien ohne die künstlichen Basenpaare zerstört. „Dadurch blieben bei der Bakterienvermehrung nahezu 100 Prozent der nichtnatürlichen Basenpaare in der DNA erhalten – und das nahezu unbegrenzt lange“, so Romesberg und seine Kollegen. Auch nach 60 Zellteilungen der semisynthetischen Bakterien gab es so gut wie keine Verluste.
„Lebewesen mit unnatürlichen Eigenschaften“
Die neue semisynthetische Mikrobe ist damit deutlich robuster und vermehrungsfähiger als ihr Vorgänger. Nach Ansicht der Wissenschaftler ist dies ein wichtiger Fortschritt. „Dieser optimierte semisynthetische Organismus liefert uns nun eine geeignete Plattform, um Lebewesen mit völlig unnatürlichen Eigenschaften und Funktionen zu schaffen, die es in der Natur nicht gibt“, konstatieren die Forscher.
Einigen dürfte angesichts solcher Begeisterung für Eingriffe in den Code des Lebens eher das Gruseln kommen. Zumal bisher völlig unklar ist, welche Risiken mit solchen und ähnlichen „Frankenstein“-Mikroben verbunden sind. Ähnlich wie bei anderen Eingriffen ins das Genom ist die Forschung hier der ethischen und rechtlichen Debatte einen Schritt voraus.
Zwar betonen die Forscher, dass ihre halbkünstlichen Mikroben in der freien Umwelt nicht lebensfähig sind, weil ihnen dort die chemischen Bausteine für ihre synthetischen Basen fehlen. Doch schon bei weiteren Änderungen an den Basen könnte dies anders aussehen. (Proceedings of the National Academy of Sciences, 2017; doi: 10.1073/pnas.1616443114)
(PNAS, 24.01.2017 – NPO)